Uehara, T., Nakamura, T., Yao, D. et al. S-Nitrosylated protein-disulphide isomerase links protein misfolding to neurodegeneration. Nature 441, 513–517 (2006).
* 958회 인용, Google Scholar 2023 Mar 8 검색 기준

2006년에 출간된 Nature Letter 분류의 article.
잡담
현재의 Nature는 6쪽 짜리냐, 8쪽 짜리냐, 두 가지 타입으로 논문을 출간하지만 2020년(?) 전까지는 6쪽 이내의 짧은 article은 letter 라는 별도 형식으로 출간했다.
Letter 형식에 실리는 논문은 아무래도 figure가 작고, 적고, 본문도 짧다. 그런 즉 짧고 강렬한 research article이 자주 실리곤 했다. 지금은 PNAS에서 많이 볼 수 있는 톡톡 튀는 아이디어를 좀더 강조했다고나 할까. 각자 본인 분야의 고전, 역사적 명작, 분야를 열어젖힌 논문이 궁금하다면 Nature Letter 분류를 뒤적여보면 좋지 않을까 한다.
분야에 따라 물리학은 Physics Review Letter 등 각자의 '명작 코너' 저널이 따로 있겠지만서도...
뇌, 특히 대뇌의 신경퇴행성 질환인 Amyotrophic lateral sclerosis (ALS), Parkinson’s disease (PD), Alzheimer’s disease (AD), Huntington’s disease (HD) 등에 세포 내 노폐물이 관련되어 있다는 사실은 오래된 추측이며, 현재 어느 정도 입증되기도 했다. 최근까지 핫하다가 잠시 주춤하고 있는 β-amyloid 가설 같은 경우도 제대로 기능하지 못하는 단백질이 뇌 세포인 뉴런에 쌓여 세포를 비정상적인 상태로 변성 시킨다는 이론에서 출발한다.
사실 신경퇴행성의 이유는 여전히 명확하지 않다. 흔히 risk factor라고 하는, '이런저런 짓을 하거나 이런저런 물질에 노출되면 위험성이 증가한다'는 정보는 많지만 '어떤 과정을 거쳐서 신경 세포의 비정상기능에 이르는가?'는 명쾌하지 않다는 뜻이다. 좀더 자세히 풀어쓰자면 '액셀을 밟으면 차가 가속한다'는 수준의 지식과 '액셀을 밟으면 자동차 내부의 부품이 어떤 순서로 작동하고 서로 간에 어떤 방식으로 힘을 전달하여 최종적으로 가속하는지' 전부 파악하는 수준의 지식은 다르다. 과학자들은 전자의 많은 부분을 파악했고, 후자에서도 많은 정보를 축적했지만, 후자의 지식을 완성하지는 못했다.
현재 2023년. 2006년 논문은 상당히 오래된 논문이지만은, 이 논문은 "어떤 단백질(PDI)이 어떤 식으로 기능을 상실하고(S-nitrosylation), 그 결과 왜(misfolded protein reponse) 세포에 어떤(malfunctioning protein degradation) 문제가 생기는지' 파악한 논문이라는 점에 의의가 있다.
*배경 지식*
배경지식부터 짚고 넘어가자면,
0. NO (일산화질소), CO (일산화탄소), H₂S (황화수소)
인체에서 자연스럽게 생기는 기체를 꼽자면, 제일 잘 알려진 CO₂를 제외하고 위의 3가지가 남는다. 각각 무엇인고 하니, 혈관 확장 등의 기능이 있지만 많이 발생하면 세포가 죽는 놈 (NO), 들이 마시면 적혈구에 산소 대신 들러붙어 질식을 일으키는 놈 (CO), 달걀 썪는 냄새의 근원 (H₂S)이다. 썩 좋아 보이는 놈은 없는데... 싶지만 세 가지 모두 각자 나름대로 세포 안에서 역할이 있다. 이번 논문은 그 중 NO를 다룬다.
1. ROS/RNS
reactive oxygen 혹은 nitrogen species의 줄임말.
산소, 혹은 질소를 포함하며 화학적 반응성이 높은 화합물의 통칭이다. 홀전자(표에서 검은 점• 으로 표시)가 있다면 특히 라디칼(radical) 이라 따로 분류한다. 보통 세포 안에 이 물질이 생기면 단백질이고 핵산이고 인지질이고 닥치는대로 산화 시켜 구조를 망가트리기 때문에 '몸에 안 좋다'는 인식이 있는데, 산화-환원 교환비가 일대일이라 분자 하나가 세포를 엉망진창으로 만드는 그런 놈은 아니다.
세포가 산소를 이용해 에너지를 만들고, 질소가 포함된 아미노산을 합성 및 분해하는 과정에서 슬금슬금 불가피하게 생기는데, 단순히 해로운 부산물이기만 하지는 않고 나름대로 역할이 있기도 하다. ROS이기도 하고 RNS이기도 한 ONOO¯는 정말 정말 강력한 산화제이며 세포 내에 쌓이면 세포 기능을 확연히 떨어뜨리는 악명 높은 물질이다. 생긴 거 답게 세포 안에 ROS와 RNS가 쌓이면 그 둘이 합쳐지면서 생긴다.

2. S-nitrosylation (SNO, S-NO)
단어를 해석하자면 S가 뜻하는 것은 '황'이고, 황에 NO라는 작용기가 붙어서 S-N-O 순으로 원자가 이어져있다는 뜻이다.
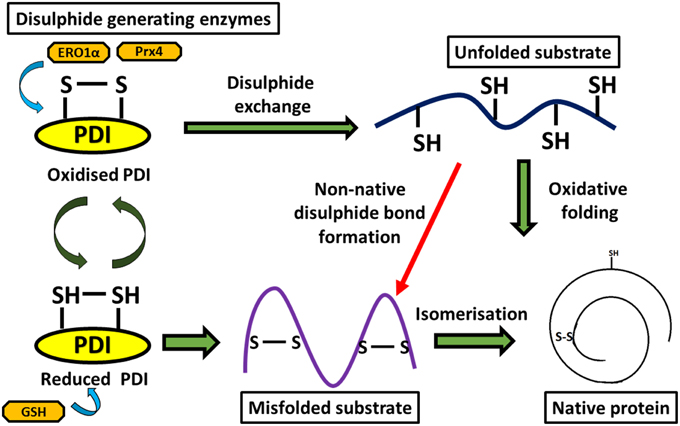
PDI 다루는 김에 PDI 리뷰 논문에서 그림을 가져오자면... 오른쪽 위의 'unfolded substrate'를 보면 –SH 라고 적힌 작대기가 삐죽 나와있다. 단백질을 이루는 원소들이 어찌저찌 복잡하게 연결되어 있는데, 그 가장 표면에 (덩어리)–S–H 순으로 원소가 이어져있다는 뜻이다. 논문을 읽으려 시도한다면 보통 이 정도 내용은 알겠지만... 혹시 몰라서 설명해둠.

단백질을 이루는 아미노산 중에 시스테인이란 녀석은 메티오닌과 함께 유이하게 황(S)을 포함하고 있고, 유일하게 –SH 작용기를 가진다. 이 작용기에는 thiol group 이라는 이름도 있다. PDI 언급하는 두 개 위 그림에서 보이듯이 thiol group 두 개를 연결해서 (덩어리1)–S–S–(덩어리2) 꼴로 만들 수 있다.
이 구조물(?)을 이황화결합, 이황화 다리 (bridge) 라고들 부르며, 튼튼한 공유결합이라 단백질의 구조를 안정화하는 데에 무척 중요하다.
그런데 이 황이 환원되어 thiol group (–SH) 형태로 비어있다면, 꼭 다른 thiol group이 아니어도 산화성 화합물이 와서 공격하기 쉽다. 단백질을 이루는 모든 시스테인의 모든 thiol group이 이황화결합에 사용되는 것도 아니고 그렇다고 또 다른 산화형으로만 존재하는 것도 아니기에... 정상적인 단백질에서는 시스테인의 thiol group이 환원된 형태로 있는 경우도 제법 있다. 그렇게 빈 자리가 있는데 그 주변에 산화성 분자가 어슬렁거리고 있다면.. 아래 그림처럼 다양한 형태로 황(정확히는 황화수소 꼴 −Sh인데, 수소 H를 떼어버리고 지가 붙는 거임)에 뭐가 들러붙는다!
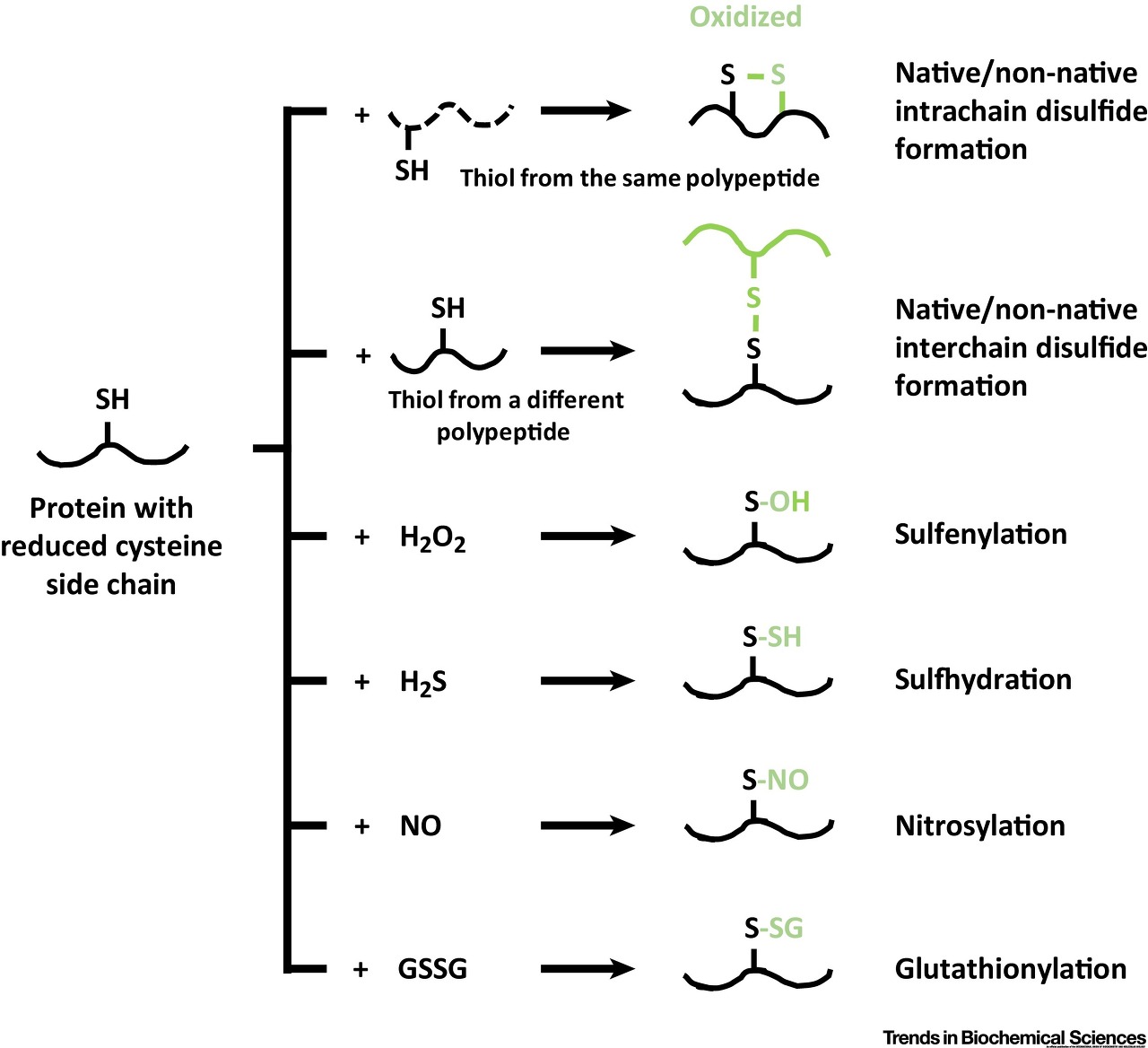
그 중에 밑에서 두 번째 황에 N과 O가 순서대로 붙은 –S–NO 꼴. 그걸 S-nitrosylation (SNO, S-NO) 부른다.
단백질을 연구하는 과학자들은 시스테인에 뭔가 들러붙는 경우가 있는지, 뭐가 어디에 들러 붙는지, 그리고 "뭔가 들러붙으면 무슨 일이 벌어지는지", 다시 말해 "단백질의 구조와 기능이 변하는지" 를 탐구했다. 그 결과 중 하나가 이번 논문이겠다.
**논문 해설**
첫 번 째 질문: PDI에 SNO modification이 생길까? 생긴다면 어느 시스테인에 생길까?
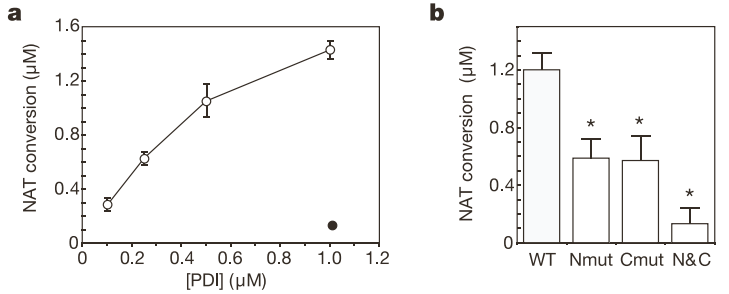
저자들은 먼저 purified PDI, 즉 세포 밖에서 만들어지고 정제된 PDI 효소에 SNOC(SNO-Cystein)를 처리하고 이 PDI가 SNO-PDI가 되는지 보았다. DAN assay를 요약하자면 이렇다. SNO 처리가 된 시스테인을 가하면 PDI에 NO기가 옮겨 붙고, DAN이란 물질도 NAT이란 형태로 변한다. FIGURE 1a는 그렇게 SNOC를 처리하거나(빈 동그라미), 처리하지 않은(검은 동그라미) PDI를 여러 농도로 녹여놓고 DAN assay로 NAT의 농도를 본다. PDI 농도가 증가할수록 NAT의 농도가 증가한다는 것은, PDI가 SNO-PDI로 바뀌고 있다는 뜻. 즉 세포 밖에서 만들어진, 정제된 PDI는 SNOC에 의해 SNO-PDI로 변한다. 이렇게 실제 생체 환경과 괴리가 있는, 통제된 실험관 환경의 실험을 in vitro라고 한다. 일반적인 생물학 논문은 처음부터 큰 스케일을 봐야하는 행동학이나 생태학 계열이 아니라면, in vitro에서 in vivo/ex vivo로 나아가며 "진짜 생명체 안에서도 그 반응이 재현돼?" 라는 질문에 대답한다는 인상을 받곤 한다.
FIGURE 1b는 PDI의 N-terminal domain과 C-terminal domain 이라고 부르는 두 영역, 그리고 그 사이 어딘가에 있는 시스테인, 합쳐서 총합 6개의 시스테인 중에 어느 게 SNO가 붙는지 본다. 6개를 하나 씩 스캐닝하지는 않고, N-terminal 쪽 2개와 C-terminal 쪽 두개를 각각 세트로 묶어버렸다. 걔네가 효소로 작동하는 구역에 있었기 때문. 구조적인 기능만 있는 거 같은 가운데 끼인 2개는 일단 무시함. 그렇게 N-terminal의 시스테인 두 개를 NO가 붙을 수 없는 세린이란 놈으로 바꿔버린 게 Nmut PDI, C-termianl에 속한 두 개의 시스테인을 마찬가지로 날려버린 게 Cmut PDI이고, 둘 다 즉 총 4개의 시스테인을 싹다 날린 게 N&C다. 보니까 한 세트를 날리면 SNO-PDI가 되는 비율이 반 줄어든다. 두 세트를 다 날려버린 N&C PDI는 SNO-PDI form으로 바뀌지 않는다. N-terminal과 C-terminal의 시스테인 세트가 SNO 화하는 비율이 엇비슷하고, 그 4개 시스테인이 SNO기가 붙는 표적인 거 같음~
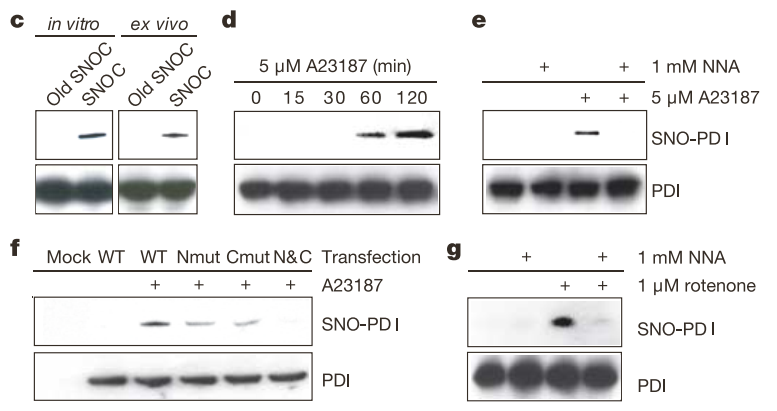
그러면 이제 실제 사람 세포에서 나온 PDI도 SNOC 처리하면 SNO-PDI가 되나 볼까? 그 질문에 답한 게 FIGURE 1c. 생명체에서 분리해낸 걸 ex vivo라고 한다. 그 다음에는 세포에 NOS를 발생시키는 약물(A23187, Ca⁺⁺ ionophore)나 nNOS 저해제(NNA)를 세포에 처리했을 때 PDI가 SNO-PDI가 되는지 봤다. 약간 의문인 것은 nNOS라는 이름에서 보듯이 이 약물은 보통 신경세포(neuron)에 작용해서 nNOS(neuronal NOS)를 만들거나 억제하는데, 막상 실험한 세포는 신장 세포주인 HEK293이다. HEK293에 A23187 처리한다고 NO가 풍부하게 생기는 게 맞는 거임? 그건 입증 안 했네? 아무튼 FIGURE 1d-e에 따르면 세포 바깥에서 만든 PDI가 아니라 실제 세포에서도 PDI는 NO가 풍부해지는 환경에서 SNO-PDI로 변한다.
FIGURE 1f에서 세포 안에 Nmut, Cmut, N&C 돌연변이를 일으켜서 확인해보니 in vitro 실험과 마찬가지로 Nmut, Cmut는 절반 정도 SNO-PDI가 되고 N&C는 SNO-PDI가 되지 않는다. 마지막으로 FIGURE 1g를 보자면, 세포에 rotenone 이란 약물을 쳐서 스트레스를 빡 주어보니(이거 살충제로도 씀)... 세포가 스트레스를 받으면 세포 안에 NO가 생긴다고들 하는데, 실제로 rotenone에 반응해서 PDI가 SNO-PDI로 변했다! 근데 rotenone과 NNA 같이 쳤다고 또 SNO-PDI가 하나도 안 생긴 건 좀 수상하지 않나? Western blot 데이터를 너무 입맛대로 편집한 거 같은데... 조작까지는 아니고 이미지의 contrast (흑백 대조 차이)를 조절해서 원하는 그림이 되게끔 좀 만진 거 같다~는 느낌이 있다. 일단 Nature 란 저널이 알아서 검증했을 거라 믿고 넘어가겠음...
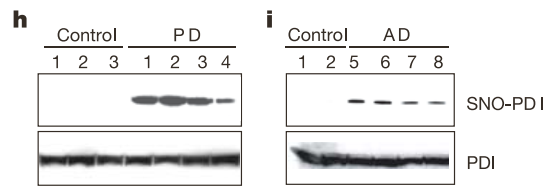
아, 근데 PDI가 SNO-PDI가 뭐랑 관련이 있는 거 같다는 게 이 논문의 발단이었지? PD 환자나 AD 환자의 신경퇴행성 질환의 이유가 세포 내에 생긴 NO 때문에 PDI가 SNO-PDI가 되고, 결국 신경 세포가 망가지는 게 아닐까, 하는 가설 때문이었지? 이제 죽은 PD, AD 환자의 뇌 조직에서 세포를 추출해서 SNO-PDI가 있는지 봤다. '어라리, 엄청 많네!' 하는 게 FIGURE 1h-i의 이야기.

그러면 어디 PDI가 SNO-PDI가 되거나 N&C 시스테인-to-세린 교체 돌연변이가 되면 세포가 진짜로 망가지는지 볼까나? 이번에도 일단 세포 바깥에서 시작한다. FIGURE 2a에서는 protein aggregation.. 그러니까 단백질이 제대로 안 만들어져서 뭉쳐진 쓰레기 덩어리가 얼마나 생기나 한번 볼까~. 이상하게 잘못 접힌 단백질을 정제해서 물에 녹이고 PDI를 넣어준 다음 제대로 풀어서 다시 접어주나 보는 식으로 PDI의 기능성을 측정할 수 있겠다. 이 잘모 접힌 단백질은 혼자 놔두면 막 뭉쳐버린다.
정상적인 PDI(파랑 네모)는 protein aggragation이 별로 생기지 않는다. 그도 그럴 게 PDI가 원래하는 일이 이상하게 잘못 '접힌' 단백질을 다시 풀어서 제대로 잘 조립해주는 역할이다. 이런 역할을 하는 단백질을 샤페론이라고 하고. 그러면 SNO-PDI(빨강 네모)는? 파랑 보다 2배 가량 protein aggregation이 많이 생긴다. PDI가 없는 그냥 물(흰색, 동그라미)이나 SNOC만 왕창 처리한 경우(녹색, 세모), N&C 돌연변이 PDI(시안)은 protein aggregation이 100% 로 생겨버린다. SNO-PDI도 활성을 완전히 잃었다면 좀더 멋진 데이터였겠지만, N-terminal이나 C-terminal 한 쪽만 SNO기가 붙어서 기능이 약간 가능했을 수도 있고, 양 쪽 다 SNO-PDI가 되어버려도 약간은 기능을 할 수도 있고? 아무튼 SNO-PDI도 PDI 대비 절반만 기능을 잃고 완전히 불활성화 되진 않네.
FIGURE 2b는 isomerase activity 라고 해서... PDI가 isomerase니까 그 기능을 본 건데 실험 기법은 모르겠음. 아무튼 이 것도 FIGURE 2a랑 마찬가지로 wild type PDI 기준으로 SNO-PDI랑 N&C(이름을 여기서부터 dnPDI로 바꿈)는 기능이 떨어진다.
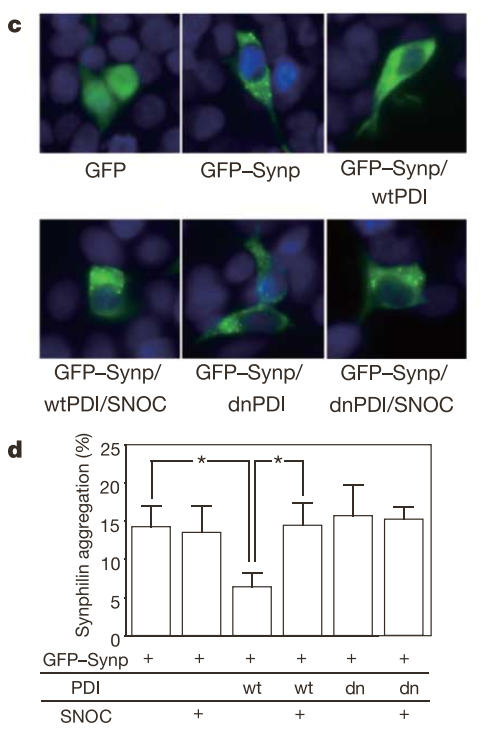
아싸, 좋았다. SNO-PDI는 PDI 보다 기능이 떨어진다! 그럼 이걸 신경세포에 넣어봐야지. 정확히는 뇌에 생긴 종양세포를 떼어서 키워낸 건데 그걸 정상 신경세포처럼 여기고 써도 되나... 사람에게서 떼어낸 조직을 배양해다가 실험실에서 키우는 거다보니 neuron의 primary cell을 쉽게 구할 수 없단 건 이해하지만서도... 그야 "멀쩡한 당신 뇌를 좀 뜯어다가 실험용으로 써도 될까요?" 할 수는 없으니깐.
그리고 세포 안에서 protein aggregation 을 보기 위해 GFP 녹색형광이 달린 Synp라는 단백질을 넣어본다. 이 단백질이 뭉치면 녹색 빛이 몽골몽골 점처럼 생긴다. GFP만 넣으면 세포 저네가 녹색으로 빛나네~ 안 뭉치고 골고루 잘 퍼졌음. GFP-Synp 는 쩜쩜쩜쩜 뭔가 뭉친 게 보임. 여기에 PDI나 PDI/SNOC(SNO-PDI 생기게), dnPDI, dnPDI/SNOC 각각 처리해볼까? 정상 PDI가 일하고 있는 세포는 쩜박이가 아니다. 그런데 PDI/SNOC는 다시 쩜박이가 되네. SNOC 처리하면 PDI가 SNO-PDI가 되니까, SNO-PDI는 protein aggregation을 막지 못한다. dnPDI도 기능은 구리다고 했고 실제로 세포 안에 일을 못한다. dnPDI/SNOC는 혹시 PDI/SNOC가 일을 못한 게 SNO-PDI가 생겨서가 아니라 SNOC 자체에 뭔가 있을까봐 넣은 컨디션이다. dnPDI나 dnPDI/SNOC나 별 차이가 없다면 SNOC 때문에 PDI/SNOC에서 protein aggregation이 증가한 건 아니겠군~ 하고 확인할 수 있다.
근데 GFP만 넣은 세포는 핵(파란색 형광 염색)이 왜 안 보이죠... 뭐임... 빈 깡통임?
위에서 보인 세포 사진을 정량까지 한 게 FIGURE 2d의 그래프이다. 요즘이야 정량하는 게 당연하지만 2006년에는 세포 사진 같은 데이터는 정량하지 않고 뭉개고 넘어가기도 해서, 상당히 공들인 논문이라고 한다 (교수님 말씀).

그러면 이런 식으로 세포 안에 몽글몽글이 생기면 무슨 일이 벌어질까? 세포도 이걸 가만히 두고 보지는 않는다. poly Ubiquitination이라고 해서 Ubiquitin을 여러 개 다닥다닥 이 aggregate에 붙인다. 세포 내부에서 쓰는 대형 폐기물 처리 스티커라고나 할까... 이걸 붙여두면 세포 내 단백질 쓰레기 청소부인 proteasome이 와서 부순다. 적어도 부수려고 시도는 한다. PDI가 정상이면 aggregation도 억제되고 poly Ubiquitination (빨강 형광)도 보이지 않는다.
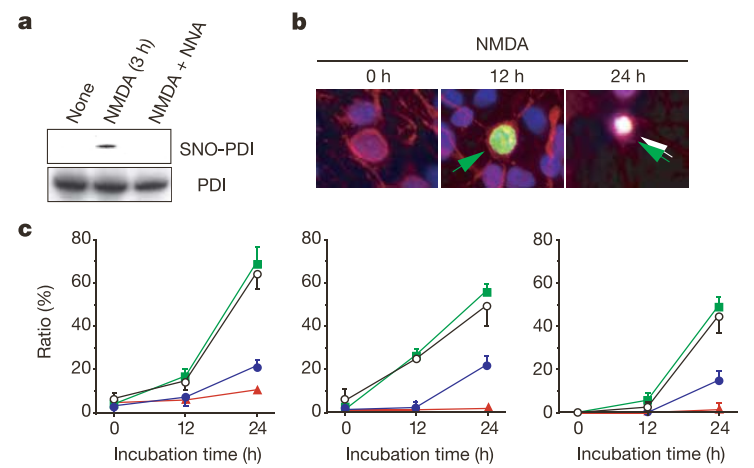
FIGURE 3에서도 신경세포 괴롭히기를 이어간다. 신경세포는 NMDA라는 세포 외부 물질에 자극을 받으면 칼슘 채널이 열려서 Ca⁺⁺ 이오니 세포 안으로 쏟아져들어온다. Ca⁺⁺ 이온이 세포 내부에 많아지는 건 세포 입장에서 굉장한 스트레스고, 여러 방어 기작을 펼치면서 그 부산물로 NO도 쌓인다. NO가 쌓이면 PDI는 또 SNO-PDI가 되어버리고 기능이 반 토막 난다. 저자들은 FIGURE 3a에서 NMDA를 치면 SNO-PDI가 확 늘어나고, NMDA에 NOS 생성을 억제하는 NNA를 같이 치면 SNO-PDI가 생기지 않는다는 걸 보였다.
세포 사진을 한번 볼까? FIGURE 3b에서 세포에 신경 세포에서만 발현하는 MAP2라는 단백질(빨강)을 관찰한다. 이 세포에 NMDA를 처리하니 12시간 후부터 poly Ubiquitination(초록)이 보인다. 아까는 Ubiquitin을 빨강으로 봤잖아, 헷갈리게 하지마. NMDA를 처리하니까 세포가 스트레스 받아서 protein aggregation이 일어난다. 그리고 24시간이 지나면 세포가 쪼그라든다. 저러면 곧 죽는다.
이번에도 FIGURE 3b를 FIGURE 3c에서 정량을 하자. 그렇게 세포가 죽거나(왼쪽), poly Ubiquitination이 활발하게 일어나거나(가운데), 둘 다 일어난 경우(오른쪽)가 전체 세포 중에 얼마나 되는지 비율을 측정해 도표로 그렸다. PDI가 정상(빨강)이면 세포가 죽지 않고 세포 내 쓰레기도 많이 생기지 않는다. 역시나 세포에 SNO-PDI나 dnPDI를 넣으면 넣어봤자 세포는 골로 간다.

그럼 어디 이번엔 protein aggregation 말고~ 세포가 NO의 발생에 스트레스를 받고 있다는 것을, 그리고 정상적인 PDI는 이 문제를 해소할 수 있지만, SNO-PDI는 그렇지 못하다는 걸 다른 방법으로 교차검증해볼까? 세포 내부에서 단백질은 소포체(ER)에서 가공되곤 한다. 여태까지 말은 안 했지만 PDI도 소포체 안에서 활동하는 효소, 샤페론이다. protein aggregation이 생기는 원인인 '이상하게 잘못 접힌 단백질'이 너무 많이 쌓이면 소포체는 과부하에 걸린다. 계속 단백질을 만들어서 가공하라고 공장에 밀어넣는데 공장은 지금 불량품 걸러내고 수리하기도 바쁘다. 이걸 고급진 말로 ER stress 라고 하며, ER stress가 발생하면 소포체는 "나는 할 만큼 했다. 이 세포는 망했다. 죽자! (아니면 문제를 해결하던지!)" 하는 신호를 내보낸다. 이 역할을 위해 XBP-1 이라는 단백질의 mRNA가 세포 내에 항상 대기를 타고 있다. 517 bp 라고 적힌 부근에 일정한 양의 XBP-1 mRNA 밴드가 뜨는 게 그 의미다.
XBP-1의 mRNA는 ER stress 발생 시 splicing을 거쳐 (processed) 단백질로 번역 가능한 형태로 완성된다. Xbp1 단백질은 Chop 이라는 다른 단백질이 만들어지게 유도하고, 세포 내 단백질 생산 공장이 파업함에 따라 세포가 죽거나 단백질 가공 과부하가 해소되어 상태를 회복하기도 한다. 뭐... 보통 죽는다. 외부에서 세포를 파괴하는 게 아니라, 이렇게 "이제 이 세포는 운명을 다했다..." 하고 자폭하는 걸 apoptosis 라고 하는데, 이것도 분야가 엄청 넓고 재밌다.

FIGURE 4a-c을 보자. 마지막 실험으로 세포에 PDI나 dnPDI를 넉넉하게 발현 시켜주고, SNOC를 넣어서 SNO-PDI도 만들어주고, 각각의 경우마다 ER stress 유발하는 약물(Thapsigargn[탑시갈진], Tunicamycin[튜니카마이신], MG132)을 처리해서 세포가 죽는지 본다. 정상 PDI 없이 ER stress 가 발생하면 세포는 50% 가량 죽는다. 정상 PDI가 있다면 20~40% 정도 죽는다. 정상 PDI를 발현해도 SNOC를 처리해서 SNO-PDI로 만들어버리면 50~60%의 세포가 자폭한다.
FIGURE 4d는 Pael-R 이라는 ER stress를 유발하기 쉬운 신경 세포 단백질을 왕창 발현해서 약물에 의해 세포가 뻗어버리게 하는 게 아니라 자연적인(?) ER stress가 생기게끔 유도한다. 저 위에 그림에서 GFP-Synp 쓴 거랑 비슷한 역할인데, 실제로 PD 환자의 세포에선 Pael-R이 많이 발견되어 질병의 원인으로 지목 받기도 한다. 환자의 뇌가 ER stress 유발 약물에 노출되는 경우 보다는 세포에 뭔가 이상이 발생해 단백질 가공에 실패하고 뇌 신경 세포가 죽는다고 보는 게 합당하다. 그러니 FIGURE 4d는 환자의 뇌에서 벌어지는 일을 흉내내보려는 시도인 셈이다. 역시나 정상 PDI가 충분하면 세포는 25% 정도 죽는다. SNO-PDI가 생기게끔 PDI/SNOC 처리를 하면 40% 까지 자폭 세포가 증가한다. 기능을 완전히 잃은 dnPDI는? 50%의 세포가 죽어나간다.

논문은 이 그림 FIGURE 4e로 막을 내린다. 일종의 '우리가 이 논문에서 밝힌 것...에 약간 양념 더했음' 형태의 도표인데, 내가 단백질구조학에서 시스템생물학으로 분야를 바꾼 탓인지 몰라도 최근 출간된 article에서는 잘 못 본 것 같다. 아마 '내가 이 연구에서 확실히 규명한 부분' 이외의 정보가 첨가되어야 이렇게 교과서에 실릴 법한 깔끔한 도식을 그리기 쉬운데, 최근 트랜드에선 '내가 만든 데이터'가 아닌 그림을 싣는 걸 지양하는 추세라 그럴지도. 논문을 충분히 많이 인용하면 괜찮을 수도~ 안되면 저널 에디터가 수정하라고 얘기할 듯~
아무튼 정리하자면 NMDA 라는 신경세포 신호물질이 과도하게 세포에 작용하면, 세포 내부에 Ca⁺⁺ 이온이 쌓이고, 그걸 해결하려다보니 NO가 왕창 생기고, NO가 PDI에 결합해 SNO-PDI 형태가 되면 신경 세포의 단백질 품질 관리 시스템이 잘 작동하지 않아서 세포가 죽는다는 이야기. 도파민 분비를 담당하는 신경세포가 죽으면 PD가 발생하곤 하므로, "NO가 세포 안에 너무 쌓이지 않게, 혹은 PDI가 SNO-PDI 형태로 변하지 않게 잘 조절하면 퇴행성 뇌질환을 예방할 수 있지 않을까?" 라고 제안하며 논문이 끝난다.
요즘이라면 임상시험을 했냐느니, 환자 샘플로 sequencing을 돌려봤냐느니, PD와 AD 환자에게 보편적인 현상인지 일부 환자에 국한된 현상인지 입증하라느니, 이게 상관관계인지 인과관계인지 밝히라느니, Western blot이 아니라 ELISA는 해봤냐느니, 엄청 많고 정교하고 비싼 실험을 요구할텐데...
그래도 나름대로 가설에서 결론까지 깔끔하게 논리를 쭉 끌고 나가는 논문이라 생각한다. 생물학 논문에 친숙하다면 완전히 다른 분야여도 읽기 편하게끔 글이 간결하면서도 정보가 풍부하더라. 굿~
***실험 기법: 수정 예정***
아래는 이 논문이 사용한 실험 기법들로, 나도 공부하려고 적어둠
DAN assay
Biotin assay